3.3. Scikit-image: 画像処理¶
著者: Emmanuelle Gouillart
scikit-image は画像処理に特化した Python 画像ライブラリで、 NumPy 配列を画像オブジェクトをネイティブに扱います。この章では scikit-image を多様な画像処理タスクにどう利用するかや NumPy や Scipy などの他の Python の科学技術モジュールとの連携についても扱います。
参考
基本的な画像操作、たとえば画像の切り抜きや単純なフィルタリングなど、多くの単純な操作は NumPy や SciPy でも実現できます Numpy と Scipy を利用した画像の操作と処理 を参照して下さい。
この章を読む前に前の章の内容について慣れておく必要があります、マスクやラベルといった基本操作は準備として必要です。
章の内容
3.3.1. 導入とコンセプト¶
画像は Numpy 配列 np.ndarray
| image: | np.ndarray |
|---|---|
| pixels: | 配列の値: |
| channels: | 配列の次元 |
| image encoding: | dtype (np.uint8, np.uint16, np.float) |
| filters: | 関数 ( |
>>> import numpy as np
>>> check = np.zeros((9, 9))
>>> check[::2, 1::2] = 1
>>> check[1::2, ::2] = 1
>>> import matplotlib.pyplot as plt
>>> plt.imshow(check, cmap='gray', interpolation='nearest')

3.3.1.1. scikit-image と SciPy エコシステム¶
scikit-image の最近のバージョンは Anaconda や Enthought Canopy のような Python 科学技術ディストリビューションのほとんどにに含まれています。Ubuntu/Debian 向けにもパッケージがあります。
>>> import skimage
>>> from skimage import data # most functions are in subpackages
ほとんどの scikit-image 関数は NumPy の ndarrays を引数にとります:
>>> camera = data.camera()
>>> camera.dtype
dtype('uint8')
>>> camera.shape
(512, 512)
>>> from skimage import restoration
>>> filtered_camera = restoration.denoise_bilateral(camera)
>>> type(filtered_camera)
<type 'numpy.ndarray'>
他の Python パッケージも画像処理に利用することができ、NumPy 配列と組み合わせて動作します:
scipy.ndimage: nd-arrays と組み合わせて。基本的なフィルタリングや数理形態学操作、領域の特性取得- Mahotas
同様にPython バインディングを持つ、強力な画像処理ライブラリ:
(ですが、それらは Python らしくなかったり NumPy と合わせて利用しづらかったり、まちまちです)
3.3.1.2. scikit-image でできること¶
Web サイト: http://scikit-image.org/
実例ギャラリー (matplotlib や scikit-learn にあるような): http://scikit-image.org/docs/stable/auto_examples/
様々な種類の関数、よくあるユーティリティ関数から高レベルな最近のアルゴリズム。
フィルタ: 画像を他の画像に変換する関数。
NumPy の仕組み
一般的なフィルタリングアルゴリズム
データ集約関数: 画像ヒストグラムの計算、極大値の位置、コーナの位置など。
他の操作: 入出力、可視化、など。
3.3.2. 入出力、データ型や色空間¶
入出力: skimage.io:
>>> from skimage import io
ファイルからの読み込み: skimage.io.imread()
>>> import os
>>> filename = os.path.join(skimage.data_dir, 'camera.png')
>>> camera = io.imread(filename)

Python Image Library でサポートされるデータ形式全てで動作します (あるいは imread の plugin キーワード引数を与えることで提供される任意の入出力プラグイン)。
URL で与えた画像パスでも動作します:
>>> logo = io.imread('http://scikit-image.org/_static/img/logo.png')
ファイルへの保存:
>>> io.imsave('local_logo.png', logo)
(imsave は PIL のような外部プラグインも利用できます)
3.3.2.1. データ型¶

画像の ndarrays 整数 (符号付き、符号なし) や浮動小数点数のどちらでも表現できます。
整数データ型ではオーバーフローに注意
>>> camera = data.camera()
>>> camera.dtype
dtype('uint8')
>>> camera_multiply = 3 * camera
異なる整数サイズを使うこともできます: 8, 16 あるいは 32バイトの符号付き、符号無し。
警告
重要(確信がもてないなら特に) skimage での 慣習: 浮動小数点数は [-1, 1] の範囲内であると仮定されます (全ての浮動小数点数で表現された画像のコントラストを比較できるように)
>>> from skimage import img_as_float
>>> camera_float = img_as_float(camera)
>>> camera.max(), camera_float.max()
(255, 1.0)
いくつかの画像処理ルーチンは動作に浮動小数点数の配列が必要なので、出力配列の型やデータ範囲は入力配列と異なることがあります。
>>> try:
... from skimage import filters
... except ImportError:
... from skimage import filter as filters
>>> camera_sobel = filters.sobel(camera)
>>> camera_sobel.max()
0.591502...
警告
上の例では scikit-image の filters サブモジュールを利用します、これは 0.10 と 0.11 の間に filter から filters に名前が変更されています、これは Python の組み込みの filter との衝突を避けるためです。
ユーテリティ関数が skimage で提供されており、dtype やデータ範囲の両方を変換します、変換は skimage の慣習に従って実施されます: util.img_as_float, util.img_as_ubyte など
詳しくは user guide を参照してください。
3.3.2.2. 色空間¶
カラー画像は (N, M, 3) または (N, M, 4) (alpha チャネルで透過度が符号化されている場合) のシェイプを持ちます:
>>> face = scipy.misc.face()
>>> face.shape
(768, 1024, 3)
異なる色空間 (RGB, HSV, LAB など。) の間の変換ルーチンは skimage.color で利用できます: color.rgb2hsv, color.lab2rgb, など。ドキュメンテーション文字列で入力画像として期待する dtype (そしてデータ範囲)を確認しましょう。
3次元画像
skimage のほとんどの関数は3次元画像を入力引数としてとることができます。ドキュメンテーション文字列で、3次元画像 (例えば MRI や CT 画像) に対して適用するとどうなるか確認しましょう。
練習問題
ディスク上のカラー画像を NumPy 配列として開いてみましょう。
画像のヒストグラムを計算する skimage 関数を探して、各カラーチャンネル毎のヒストグラムをプロットしましょう
画像をグレースケースに変換してヒストグラムをプロットしましょう
3.3.3. 画像の前処理 / 補正¶
ゴール: ノイズ除去、特徴(エッジ)抽出、...
3.3.3.1. 局所フィルタ¶
局所フィルタは、対象ピクセルの隣接するピクセルの値に応じて値を置き換えます。関数には線形な関数と非線形な関数があります。
隣接: 正方形(サイズを選択した)、円形、またはより複雑な 構造要素 。

例: 水平方向 Sobel フィルタ:
>>> text = data.text()
>>> hsobel_text = filters.hsobel(text)
以下の線形カーネルを利用して水平方向の勾配を計算します:
1 2 1
0 0 0
-1 -2 -1

3.3.3.2. 非局所フィルタ¶
非局所フィルタは画像の広い領域(もしくは画像全体)を利用して、1つのピクセルを変換します:
>>> from skimage import exposure
>>> camera = data.camera()
>>> camera_equalized = exposure.equalize_hist(camera)
広い範囲でコントラストがほぼ一様な領域のコントラストを強調します。

3.3.3.3. 数理形態学¶
数理形態学の導入のために wikipedia を参照しましょう。
画像の中から単純な形状 (構造要素) を検出し、形状が局所的に沿うか沿わないかに応じて画像を変更する
デフォルトの構造要素: ピクセルに対する4つの接続:
>>> from skimage import morphology
>>> morphology.diamond(1)
array([[0, 1, 0],
[1, 1, 1],
[0, 1, 0]], dtype=uint8)
Erosion = 最小フィルタ。ピクセルを構造要素を覆うピクセル最小値で置き換える。:
>>> a = np.zeros((7,7), dtype=np.int)
>>> a[1:6, 2:5] = 1
>>> a
array([[0, 0, 0, 0, 0, 0, 0],
[0, 0, 1, 1, 1, 0, 0],
[0, 0, 1, 1, 1, 0, 0],
[0, 0, 1, 1, 1, 0, 0],
[0, 0, 1, 1, 1, 0, 0],
[0, 0, 1, 1, 1, 0, 0],
[0, 0, 0, 0, 0, 0, 0]])
>>> morphology.binary_erosion(a, morphology.diamond(1)).astype(np.uint8)
array([[0, 0, 0, 0, 0, 0, 0],
[0, 0, 0, 0, 0, 0, 0],
[0, 0, 0, 1, 0, 0, 0],
[0, 0, 0, 1, 0, 0, 0],
[0, 0, 0, 1, 0, 0, 0],
[0, 0, 0, 0, 0, 0, 0],
[0, 0, 0, 0, 0, 0, 0]], dtype=uint8)
>>> #Erosion removes objects smaller than the structure
>>> morphology.binary_erosion(a, morphology.diamond(2)).astype(np.uint8)
array([[0, 0, 0, 0, 0, 0, 0],
[0, 0, 0, 0, 0, 0, 0],
[0, 0, 0, 0, 0, 0, 0],
[0, 0, 0, 0, 0, 0, 0],
[0, 0, 0, 0, 0, 0, 0],
[0, 0, 0, 0, 0, 0, 0],
[0, 0, 0, 0, 0, 0, 0]], dtype=uint8)
Dilation: 最大フィルタ:
>>> a = np.zeros((5, 5))
>>> a[2, 2] = 1
>>> a
array([[ 0., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 0.],
[ 0., 0., 1., 0., 0.],
[ 0., 0., 0., 0., 0.],
[ 0., 0., 0., 0., 0.]])
>>> morphology.binary_dilation(a, morphology.diamond(1)).astype(np.uint8)
array([[0, 0, 0, 0, 0],
[0, 0, 1, 0, 0],
[0, 1, 1, 1, 0],
[0, 0, 1, 0, 0],
[0, 0, 0, 0, 0]], dtype=uint8)
Opening: erosion + dilation:
>>> a = np.zeros((5,5), dtype=np.int)
>>> a[1:4, 1:4] = 1; a[4, 4] = 1
>>> a
array([[0, 0, 0, 0, 0],
[0, 1, 1, 1, 0],
[0, 1, 1, 1, 0],
[0, 1, 1, 1, 0],
[0, 0, 0, 0, 1]])
>>> morphology.binary_opening(a, morphology.diamond(1)).astype(np.uint8)
array([[0, 0, 0, 0, 0],
[0, 0, 1, 0, 0],
[0, 1, 1, 1, 0],
[0, 0, 1, 0, 0],
[0, 0, 0, 0, 0]], dtype=uint8)
Opening は小さなオブジェクトを取り除き、コーナーをスムーズにします。
グレースケールに対する数理形態学
数理形態学の演算は(2値でない)グレースケール画像(整数や浮動小数点数)に対しても利用できます。erosion や dilation は最小(同様に最大)フィルタに対応します。
より高度な数理形態学操作が利用できます: tophat, skeletonization など。
参考
基本的な数理形態学演算は scipy.ndimage.morphology にも実装されています。 scipy.ndimage は任意次元で動作するように実装されています。
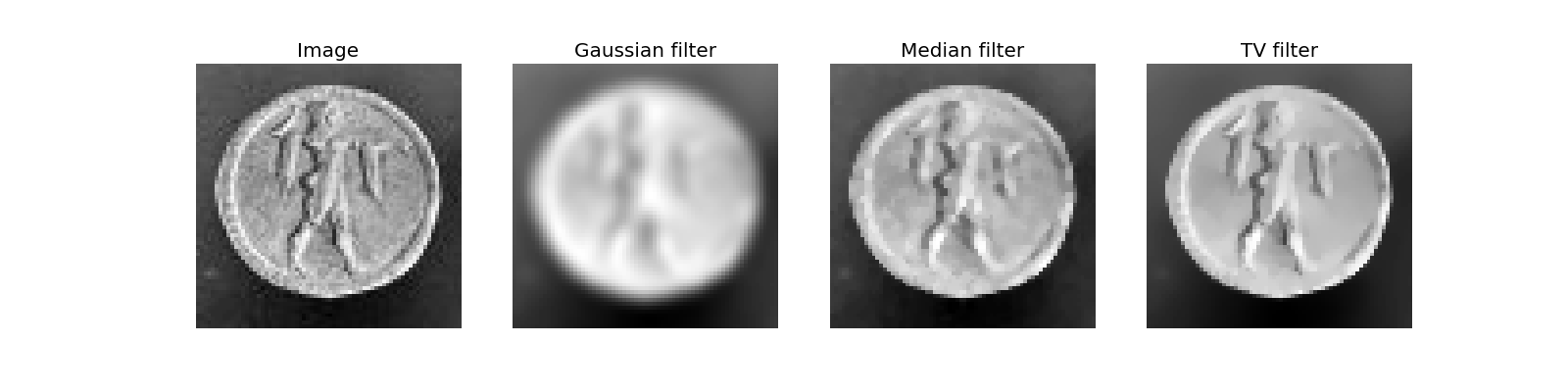
フィルタ比較の例: 画像のノイズ除去
>>> from skimage.morphology import disk
>>> coins = data.coins()
>>> coins_zoom = coins[10:80, 300:370]
>>> median_coins = filters.rank.median(coins_zoom, disk(1))
>>> from skimage import restoration
>>> tv_coins = restoration.denoise_tv_chambolle(coins_zoom, weight=0.1)
>>> gaussian_coins = filters.gaussian_filter(coins, sigma=2)
3.3.4. 画像の分割¶
画像の分割とは画像の異なる領域に異なるラベルづけをすることで、例えば対象となる複数のピクセルを抽出する際に使います。
3.3.4.1. 2値分割: 前面 + 背面¶
3.3.4.1.1. ヒストグラムに基づく方法: 大津のしきい値選定¶
ちなみに
Otsu method は前面と背景を分けるための閾値を見つけるための簡単な発見的方法です。
from skimage import data
from skimage import filters
camera = data.camera()
val = filters.threshold_otsu(camera)
mask = camera < val

3.3.4.1.2. 散らばった画像のつながった要素をラベルづけする¶
ちなみに
一旦前面にあるオブジェクトを分割してしまえば、それぞれを別のものとして分けることができます。それぞれに異なる整数値のラベルを代入して分けることができます。
人工データ:
>>> n = 20
>>> l = 256
>>> im = np.zeros((l, l))
>>> points = l * np.random.random((2, n ** 2))
>>> im[(points[0]).astype(np.int), (points[1]).astype(np.int)] = 1
>>> im = filters.gaussian_filter(im, sigma=l / (4. * n))
>>> blobs = im > im.mean()
全てのつながっている要素をラベルづけする:
>>> from skimage import measure
>>> all_labels = measure.label(blobs)
前面のつながっている要素のみをラベルづけする:
>>> blobs_labels = measure.label(blobs, background=0)

参考
scipy.ndimage.find_objects() は画像内のオブジェクトのスライスを返すので便利です。
3.3.4.2. マーカーに基づく方法¶
領域内にマーカーをつけられれば、それを利用して領域を分割できます。
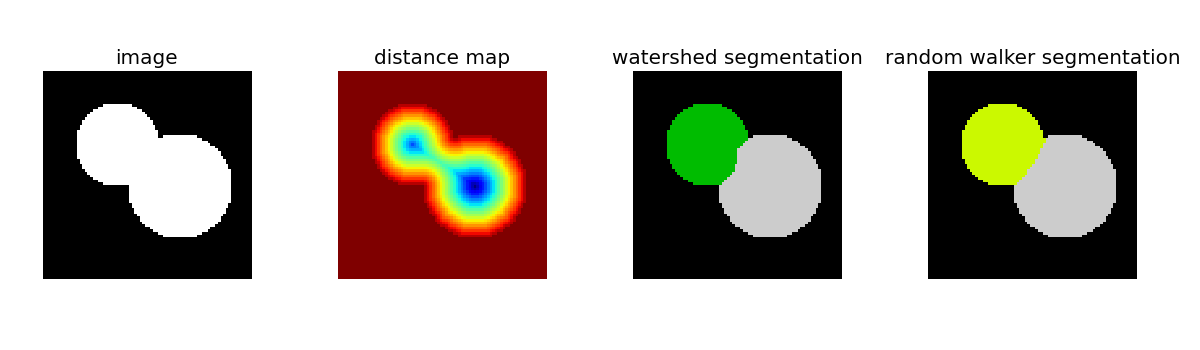
3.3.4.2.1. 分水嶺 分割¶
分水嶺アルゴリズム (skimage.morphology.watershed()) は画像の中の「たらい」をいっぱいにするように領域を広げていくアプローチです:
>>> from skimage.morphology import watershed
>>> from skimage.feature import peak_local_max
>>>
>>> # Generate an initial image with two overlapping circles
>>> x, y = np.indices((80, 80))
>>> x1, y1, x2, y2 = 28, 28, 44, 52
>>> r1, r2 = 16, 20
>>> mask_circle1 = (x - x1) ** 2 + (y - y1) ** 2 < r1 ** 2
>>> mask_circle2 = (x - x2) ** 2 + (y - y2) ** 2 < r2 ** 2
>>> image = np.logical_or(mask_circle1, mask_circle2)
>>> # Now we want to separate the two objects in image
>>> # Generate the markers as local maxima of the distance
>>> # to the background
>>> from scipy import ndimage
>>> distance = ndimage.distance_transform_edt(image)
>>> local_maxi = peak_local_max(distance, indices=False, footprint=np.ones((3, 3)), labels=image)
>>> markers = morphology.label(local_maxi)
>>> labels_ws = watershed(-distance, markers, mask=image)
3.3.4.2.2. ランダムウォーク 分割¶
ランダムウォークアルゴリズム (skimage.segmentation.random_walker()) は分水嶺アルゴリズムに似ていますが、より確率的なアプローチです。画像のラベルが拡散させるというアイデアに基づいています:
>>> from skimage import segmentation
>>> # Transform markers image so that 0-valued pixels are to
>>> # be labelled, and -1-valued pixels represent background
>>> markers[~image] = -1
>>> labels_rw = segmentation.random_walker(image, markers)
ラベル画像の前処理
skimage のいくつかのユーティリティ関数はラベル画像(つまり、異なる領域とみなせる散らばった値を持つ画像) に対して使うころができます。関数は名前で役割がわかるように命名されています: skimage.segmentation.clear_border(), skimage.segmentation.relabel_from_one(), skimage.morphology.remove_small_objects(), など.
練習問題
dataサブモジュールからcoins画像を読み込みます。いくつかの分割方法をテストして背景からコインを分けましょう: 大津のしきい値選定、適応しきい値選定、分水嶺やランダムウォーク分割。
必要なら、前処理関数を使ってコインと背景の分割を改善してみましょう。
3.3.5. 領域の特性を測定する¶
>>> from skimage import measure
例: 2つに分割された領域のサイズと周囲の長さを計算:
>>> properties = measure.regionprops(labels_rw)
>>> [prop.area for prop in properties]
[770.0, 1168.0]
>>> [prop.perimeter for prop in properties]
[100.91..., 126.81...]
参考
いくつかの特性値を取得するために異なる API として scipy.ndimage.measurements にある関数が利用できます (一覧もみられます)。
練習問題 (続き)
前の練習問題のコインと背景の2値画像を利用します。
異なるコインをラベルづけしましょう。
全てのコインのサイズと離心率を計算しましょう。
3.3.6. データの可視化と対話操作¶
有効な可視化はパイプライン処理をテストするのに便利です。
いくつかの画像処理操作:
>>> coins = data.coins()
>>> mask = coins > filters.threshold_otsu(coins)
>>> clean_border = segmentation.clear_border(mask)
2値の結果を可視化する:
>>> plt.figure()
<matplotlib.figure.Figure object at 0x...>
>>> plt.imshow(clean_border, cmap='gray')
<matplotlib.image.AxesImage object at 0x...>
等高線を可視化する:
>>> plt.figure()
<matplotlib.figure.Figure object at 0x...>
>>> plt.imshow(coins, cmap='gray')
<matplotlib.image.AxesImage object at 0x...>
>>> plt.contour(clean_border, [0.5])
<matplotlib.contour.QuadContourSet ...>
skimage の特化したユーティリティ関数を利用する:
>>> coins_edges = segmentation.mark_boundaries(coins, clean_border.astype(np.int))

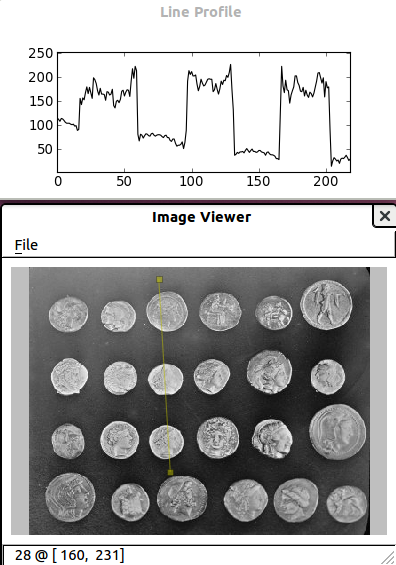
(実験的な) scikit-image ビューア
skimage.viewer = matplotlib ベースの画像表示キャンバス + 実験的な Qt ベースの GUI ツールキット
>>> from skimage import viewer
>>> new_viewer = viewer.ImageViewer(coins)
>>> new_viewer.show()
ピクセル値の表示に便利です。
より多くの操作するために、ビューアにプラグインを追加できます:
>>> new_viewer = viewer.ImageViewer(coins)
>>> from skimage.viewer.plugins import lineprofile
>>> new_viewer += lineprofile.LineProfile()
>>> new_viewer.show()
3.3.7. コンピュータビジョンのための特徴抽出¶
幾何的な特徴や表面の質感などの特徴を画像から抽出します、以下のために
画像の一部を分類する (例えば空とビル)
異なる画像の一部をマッチさせる (例えば物体認識)
さらに多くの Computer Vision の応用があります。
>>> from skimage import feature
例: Harris 検出器を利用してコーナーを検出します:
from skimage.feature import corner_harris, corner_subpix, corner_peaks
from skimage.transform import warp, AffineTransform
tform = AffineTransform(scale=(1.3, 1.1), rotation=1, shear=0.7,
translation=(210, 50))
image = warp(data.checkerboard(), tform.inverse, output_shape=(350, 350))
coords = corner_peaks(corner_harris(image), min_distance=5)
coords_subpix = corner_subpix(image, coords, window_size=13)

(この例は scikit-image の plot_corner 例から採用しています)
面白いことに、このようなコーナーは異なる画像での物体のマッチに使うことができます、scikit-image の例 plot_matching にあるように。
